
(図1) この日記で書きたかったことを漫画にしました。
TL;DR
- DNA の塩基配列は ATGC の 4 文字からなる文字列で表現できます。
- 二本鎖 DNA や二本鎖 RNA を 1 次元配列で表現できるのは、塩基対によって相補鎖を求められるため。
- DNA や RNA について調べるときに文字列の問題として計算機上で扱えば、文字列のアルゴリズムを適用できます。
NOTE: どうして専門分野外の DNA の話を?
文字列アルゴリズムに関する論文やテキストを読むと、かならずと言っていいほど「DNA は ATGC からなる文字列で表せる」と書いています。 特に文字列の編集距離の話でよく登場します。 例えば、 Text Algorithms [MR] や Algorithms on Strings [MHL] といった書籍にも出てきますし、 O(ND) Difference Algorithm [Myers] のイントロダクションでも DNA の話は出てきます。 DNA だけでなく RNA や タンパク質のアミノ酸配列の話も出てきます。 ところで、 DNA の二重らせんモデルの絵を思い浮かべてみると「どうして二重らせん構造なのに一次元配列 1本 で表せるのか」という疑問が湧き上がりました。 そこで、必要最小限の内容にしぼって「どうして DNA は文字列で表せるのか?」という話を日記に書きます。
計算機で DNA 塩基配列を扱おう
DNA や RNA, タンパク質 という言葉を聞くと生物学や化学の世界を最初に思い浮かべますが、計算機の上でそれらを表現できるのであれば計算機科学の世界の話として扱えます。 実は、計算機科学ではこれらを文字列として扱うことができます。 ただの文字列であれば、それがウイルスの進化系統樹の作成であろうと、もっと一般的な文字列の問題として扱うことができ、文字列のアルゴリズムを適用できます。 それでは何をどうやって文字列として扱っているのでしょうか?
DNA の塩基配列を文字列で表す
DNA (deoxyribonucleic acid, デオキシリボ核酸) の塩基配列は、ATGC の 4 文字からなる文字列 (または 1 次元配列) で表現できます。 例えば、実際の Variola virus (天然痘ウイルス) の DNA 塩基配列は次のように表現できます1。
CTCGAGAGTATATGTTGTTGAACGTTATTGTTTGAGAAATAGTTGATGCATCAGAATGGTTTGCATTTAT
DNA は ヌクレオチド が鎖状に繋がってできています。 ヌクレオチドは リン酸 と 糖 2、そして 4 種類の 塩基 のうちの 1 個から構成される物質です。(図2, 図3参照) 4 種類の塩基は次の通りです。
- Adenine (アデニン)
- Thymine (チミン)
- Guanine (グアニン)
- Cytosine (シトシン)
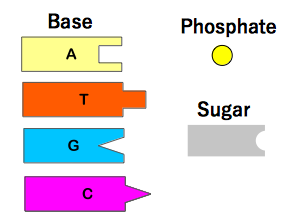
(図2) リン酸 (phosphate) と糖 (sugar) そして 4 種類の塩基 (Base) Adenine, Thymine, Guanine, Cytosine のモデル図
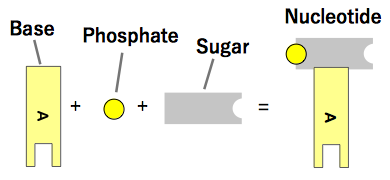
(図3) ヌクレオチド (Nucleotide) のモデル図
ATGC の文字集合は、この 4 種類の塩基 Adenine, Thymine, Guanine そして Cytosine の頭文字に由来します。 また、ヌクレオチドが鎖のように繋がったものを 塩基配列 (sequence) といいます。 DNA は 2 本のヌクレオチドの鎖が 二重らせん構造 になるように、ヌクレオチドの塩基と塩基が組み合わさってできています。(図4参照)
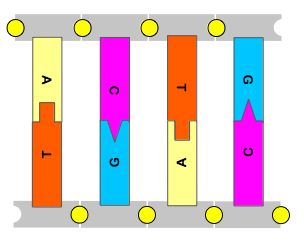
(図4) ヌクレオチドの鎖が 2 本組み合わさってできた DNA のモデル図
ここで気になるのが、どうして二重らせん構造なのに DNA の塩基配列を 1 次元配列(または文字列)で表現できるのかということです。例えば 図4 では ACTG と TGAC の 2 つの 1 次元配列を用意する必要があるのではないかと感じます。
この疑問に答えるのが 塩基対 (base pair, bp) です。A は T と、 G は C と必ず対になります。(図5参照) この組み合わせを塩基対といいます。
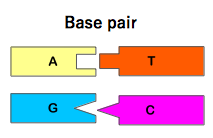
(図5) Adenine は Thymine と、 Guanine は Cytosine と塩基対を作る
DNA は二本の鎖が塩基対により 相補的 (complementary) に組み合わさってできています 3。
塩基対の関係を使えば、片方の塩基配列から対になるもう片方の塩基配列(これを 相補鎖 といいます)がわかります。例えば ACTG なら塩基対の関係から、もう片方の塩基配列は TGAC とわかります。(図6参照)
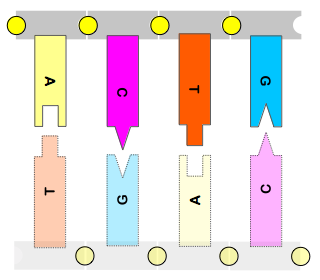
(図6) 塩基対の関係からもう片方の塩基配列を求められる
ここから DNA の塩基配列は ATGC からなる文字列(または 1 次元配列)で表現できることがわかります。
RNA
RNA (ribonucleic acid, リボ核酸) も DNA と同様に文字列で表現できます。 DNA と異なるところは、構成する塩基です 4。 RNA は次の 4 種類の塩基から構成されています。
- Adenine (アデニン)
- Uracil (ウラシル)
- Guanine (グアニン)
- Cytosine (シトシン)
また、RNA には二重らせん構造の RNA と、一本の鎖から成り立つ RNA の二種類があります5。 RNA の場合も DNA と同じように塩基対の関係があり、 A は U と、 G は C と結合します。 なので、一本鎖 RNA も二本鎖 RNA も AUGC の 4 種類の文字からなる文字列で表現できます。
実際の RNA ウイルスを文字列で表現した例を示します。 2016 年にノースカロライナで見つかった豚インフルエンザウイルスの RNA の一部です6。
ATGAAGGCAGCACTAGCAGTCCTGCTATATGCATTTACAACTGCAAATGCCGACACATTATGTATAGGCT
ここで U (Uracil) ではなく T で表記されていることに違和感を覚えるかもしれません。 RNA なので T ではなく U と表記してよいのですが、ここでは FASTA フォーマット7 の慣例に従い Uracil を T で表記しています。
タンパク質
DNA や RNA といった 核酸 と同じように タンパク質 の アミノ酸配列 も文字列で表現できます。 今回はアミノ酸配列についての説明を省略しますが、タンパク質は 20 種類のアミノ酸が 1 次元配列でつながったものです。 20種類のタンパク質はそれぞれ ACDEFGHIKLMNPQRSTVWY のアルファベット一文字で表記されます。 ちなみに、ここで使われていないアルファベットは BJOUXZ の 6 文字です。
文字列として見たときの DNA
DNA ウイルスだけでなく、地球上のどんな生物の DNA も ATGC からなる文字列として表現できます。 また、進化的に近い種は DNA の塩基配列も似ています。
DNA を文字列と見立てたとき、文字列同士の 類似度のようなもの を求めれば、進化的に近い種かどうかを調べることができます。 この類似度のようなものを計算機科学の世界では、 文字列の 編集距離 (edit distance) といいます。 編集距離にはいくつか種類があり、例をあげると レーベンシュタイン距離 (Levenshtein distance) や ハミング距離 (Hamming distance), Jaro-Winkler distance などがあります。 この中でよく使われるのはレーベンシュタイン距離です。
また多くの生物で共通する文字の並びがわかれば、生命にとって重要な塩基配列やアミノ酸配列がわかります。 この共通する文字の並びを 共通部分列 (common subsequence) と言います 8。 2 つの文字列間で共通する 部分列 (subsequence) の中で最長となるものを 最長共通部分列 (longest common subsequence, LCS) といい、計算機科学の世界では LCS を求める問題を LCS Problem といいます9。
アラインメント
遺伝子工学の世界では、共通部分列を求めることを アラインメント (alignment) と言います。 アラインメントは、2 つの文字列を比較して共通する部分を揃えて、2 つの文字列間の共通点を可視化したものです。
例えば、アラインメント前の 2 つの文字列を x, y とします。
x = "AGCTCGAATATGC"
y = "ACTGAAGAGC"
x, y のアラインメント結果は次のようになります:
AGCTCGAAT-ATGC // x
A-CT-GAA-GA-GC // y
ここで揃えるために挿入した - (ハイフン) のことを ギャップ (gap) または ホール (hole) といいます。
マルチプルアラインメント
2 つ以上の文字列の共通点を可視化したものを マルチプルアライメント (multiple alignment) (または Multiple Sequence Alignment, MSA) といいます。
マルチプルアラインメントの入力例は次のように、複数の文字列になります。 ここでは文字列 x, y, z, w を入力としてマルチプルアライメントを求めてみます。
x = "AGCTCGAATATGC"
y = "ACTGAAGAGC"
z = "CGAAGAC"
w = "TCAATT"
これらの文字列のアラインメント結果は次のようになります。
AGCTCGAAT-ATGC // x
A-CT-GAA-GA-GC // y
--C--GAA-GA--C // z
---TC-AAT--T-- // w
このマルチプルアライメントの結果から何がわかるのでしょうか? 例えば、 x, y, z, w がそれぞれヒト、牛、魚、カブトムシの DNA だとします。 すると次のことが見えてきます。
- ヒトと牛が(魚やカブトムシに比べて)近い種かも?
AAは地球上の生物にとって大事な塩基配列かも?
例では 4 つの文字列のみですが、このアラインメントの操作をたくさん集めた塩基配列のサンプルにたいして行って、インフルエンザウイルスの進化系統樹を作成したり、動物にとって重要な塩基配列やアミノ酸配列を見つけることができます。
文字列のアルゴリズムを使おう
昔はこれらの塩基配列のアラインメントを、生物学者みずから手作業で求めていたそうです。 インフルエンザウイルスの RNA 塩基配列は短いものでも 800 文字、長いと 2200 文字ほどになります10。 これを経験とセンスだけでアラインメントするのはとても大変です。 そこで DNA は文字列なので、今では計算機上でアラインメントを自動的に求められます。もちろん文字列なので文字列のアルゴリズムが使えます。
アラインメントを「文字列 x を文字列 y に変換する編集操作の手順」と見なせば、アラインメントを求める問題は 「M 個の文字列間の LCS を求める問題」または「Edit graph (エディットグラフ) を求める問題」に変換することができます。
ちなみに、以下の問題は Edit graph を求める問題に変換できます。
- 編集距離を求める (Edit distance problem)
- 最長共通部分列を求める (LCS problem)
- 文字列の差分 diff を求める (文字列 x を文字列 y にする最小の編集操作の手順)
大雑把に言ってしまうと、 Edit graph さえ求めてしまえば、編集距離も LCS も diff もアラインメントも求まります。 これらの文字列アルゴリズムの問題例に DNA の塩基配列が使われるのはそういった背景があります。
機会があれば、次回は計算機科学の世界に戻って LCS の話をします。
参考文献
- [Myers] Myers, E.W. "An O(ND) difference algorithm and its variations." Algorithmica Volume 1, Issue 1-4. (1986) 251-266.
- [MR] Crochemore, Maxime and Wojciech Rytter. Text Algorithms. (ISBN-13: 978-0195086096)
- [MHL] Crochemore, Maxime, Christophe Hancart and Thierry Lecroq. "Alighnment". Algorithms on Strings. (ISBN-13: 978-1107670990) Chapter 7.
- 星田昌紀, 遺伝子情報処理への挑戦 - コンピュータとバイオのフュージョン (ISBN-13: 978-4320026933) (1994)
- Molecular Biology of the Cell, 5th edition. - MBoC の最新版は第6版です。
- https://www.ncbi.nlm.nih.gov/genome - NCBI のデータベース。 DNA と RNA の FASTA 表記の実例として引用しました。
-
Variola virus の塩基配列の一部。NCBI Reference Sequence: NC_001611.1 から引用しました (https://www.ncbi.nlm.nih.gov/nuccore/9627521?report=fasta) ↩
-
DNA を構成する糖はデオキシリボース (deoxyribose)、RNA を構成する糖はリボース (ribose) です。 ↩
-
DNA の二本の鎖にはそれぞれ名前がついていて、発見者名をとって片方を Watson strand (ワトソン鎖)、もう片方を Crick strand (クリック鎖) と呼びます。 ↩
-
構成している糖も DNA と RNA で異なります。 ↩
-
少し前まで RNA は一本鎖だと言われていましたが、1998 年に RNAi とともに 二本鎖 RNA が発見されました。そのため、それ以前の書籍を見ると RNA に関して「一本鎖」と記述されていることがあります。 ↩
-
Influenza A virus の塩基配列の一部。NCBI GenBank: KU598329.1 から引用しました (https://www.ncbi.nlm.nih.gov/nuccore/992420281?report=fasta) ↩
-
FASTA フォーマットは広く使われている塩基配列やアミノ酸配列のデータ表現のひとつです。 FASTA フォーマットについてはこちらを参照ください: Formatting your Submission - The GenBank Submissions Handbook ↩
-
ここでは、部分列 (subsequence) と部分文字列 (substring) を区別しています。 ↩
-
実のところ LCS を求めることとレーベンシュタイン距離を求めることはまったく同じ問題です。 ↩
-
NCBI に登録されている Influenza A virus がそのくらいの長さ。長さの単位は bp (base pair) で、ATGC の 1 文字が 1 bp に相当します。 ↩
Leave a Reply